第二节 转基因小鼠的制备
在研究心血管疾病时,主要运用显微注射法、基因打靶或基因剔除等方法制备各种TGM模型。
1.显微注射法
Gordon等人首次成功地将含有HSV和SV40DNA片段的重组质粒DNA以显微注射法导入小鼠受精卵的雄原核内,得到了带有这种外源DNA顺序的TGM。1982年Palmiter等运用此法得到的所谓“超级巨鼠”(supermouse)曾引起整个生物学界的轰动。这一方法外源DNA整合率高、容量大,DNA长到50kb仍然有效,实验周期缩短,因而应用较广泛,是制备TGM的一种常用方法。
这一方法的实验程序如下:⑴准备假孕母鼠(养母):将可育雌鼠与输精管结扎后绝育的雄鼠交配,剌激雌鼠发生一系列妊娠变化而得到假孕母鼠作为受精卵转基因后的养母;⑵受精卵的准备:可育雌鼠注射孕马血清与绒毛膜促性腺激素(HCG)促使超排卵。处理后与可育雄鼠交配。次日从输卵管内收集受精卵备用;⑶基因导入:用显微注射装置将目的基因溶液导入受精卵雄性原核内;⑷胚胎移植:将已转入基因的受精卵自背部植入假孕母鼠的输卵管内,使胚胎在养母体内发育成熟;⑸对幼鼠的鉴定:①幼鼠发生断乳后自尾部提取DNA,与目的基因探针作分子杂交,鉴定外源基因是否整合,有整合的鼠称为首建鼠(founder);②建立鼠系,将带有外源基因的小鼠与未经转基因的小鼠交配、传代后,后代有50%机率带有整合的基因供实验用。也可将合适的组织进行细胞培养建立细胞系;③自小鼠内脏提取RNA,与目的基因探针做分子杂交,比鉴定外源基因的表达和表达的组织特异性。表达产物可以测定活性的,也可直接自血液或组织测定活性蛋白质,常用的方法如酶联免疫吸附试验(ELISA)或放射免疫测定(RIA)等。亦可取胚胎进行分析。显微注射法简化的实验过程如图23-2所示。我国已有少数实验室(如中国医学科学院基础医学研究所)应用此法进行载脂蛋白TGM研究。

图23-2 显微注射法制备转基因小鼠的程序
2.基因打靶与基因剔除技术
ES细胞是从哺乳动物早期胚胎发育产生的内胚团(inner,cell mass)中分离出来的。它本身是二倍体,能在体外培养,具有高度的全能性,可以形成包括生殖细胞在内的所有组织,并且在不同的培养条件下表现出不同的功能状态。这种细胞有两个特点,一是它本身可以分裂、增殖,形成细胞集落;另一特点是经过发育可以形成正常的动物后代。因此,借用ES细胞系可将人们企望的某种不完整的、无功能基因直接引入到ES细胞中,通过细胞增殖、筛选可得到丧失了某种基因功能的动物后代。正是由于ES细胞的研究成功与广泛应用,才使得基因打靶与基因剔除技术在转基因动物中的应用成为可能而且近来取得长足发展。
基因打靶是指外源DNA片段上带有与受体细胞染色体相应部位的同源序列,导入ES细胞后在同源部位发生定点整合,这种整合是通过同源重组的方式来实现的。Piedrahita等(1992)应用此技术已成功地筛选出携带突变ApoE基因杂交鼠后代。该方法的一般策略如下:

如果外源DNA含有突变失活的基因,定点整合人ES细胞染色体后取代原来的同源序列,即是基因剔除。将这样的ES细胞转入小鼠胚胎,便可得到基因剔除的基因小鼠。多采用突变基因(mutated gene)剔除正常基因以产生定位突变(target mutation)。即首先选定需要突变基因的全部或部分DNA序列,通过插入、修饰、删除或置换等手段落使其突变,成为靶载体,将靶载体导入小鼠ES细胞中,使之与细胞染色体内同源的靶序列进行重组,达到定位突变细胞内该基因的目的。然后在细胞水平和分子水平筛选富集发生突变细胞,并注射到小鼠囊胚腔内,将这些囊胚导入假孕母鼠子宫中,所产生的子代雄性嵌合鼠与正常雌鼠交配可获得生殖系携带该突变基因的纯合鼠。最后对纯合鼠的表型、基因型、基因表达及其产生等进行分析。
目前筛选真正发生了同源重组的ES细胞的方案有数种,如正负双向选择法(positive and negativeselection,PNS)、标记基因的特异位点表达法及PCR法,其中用得最多的方法首推PNS法。其基本原理是:构建含有一段与靶基因同源序列(10~15kb)的载体,该序列的一个外显子插有neor基因作为正选择的标志,在此序列3’端插有不含启动子的疱疹病毒胸苷激酶(HSV-tk)基因作为负选择,HSV-tk基因由邻近的neor基因启动子调节。用电转移(electroporation)法将重组体导入ES细胞,继续作体外培养,并以新霉素(G418)和致死核苷类似物(GANC)作双重筛选。因随机插入的DNA通常以从头至尾整合入受体细胞DNA中,HSV-tk+基因产物可使GANC转变成一种有毒物质使细胞死亡。如果发生同源重组,由于tk基因在同源区之外不能整合,neor基因在同源区之内得以保留,这样受体细胞抗G418有正选择作用,对GANC无转变功能而有负选择作用,因此认为存活的细胞是与导入基因发生同源重组的(图23-3)。Plump等(1992)用基因剔除技术已培育出缺ApoE基因的TGM模型,并利用这一AS小鼠模型研究了饮食和遗传因素对AS的影响。
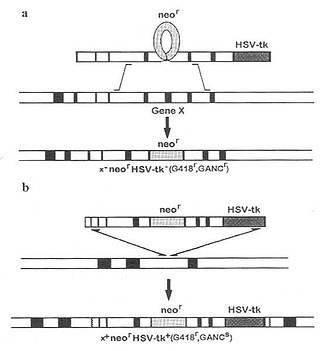
图 23-3 正负双向选择法示意图
a:同源重组;b:随机整合;neor:新霉素抗性基因;HSV-tkc:疱
疹病毒胸苷激酶基因;GeneX:干细胞内源基因(与导入基因同源);
G418:新霉素;GANC:抗致死的核苷类似物GANC;GANC:核苷类似
物参与合成,细胞致死。
近几来,随着对基因失活方面研究的深入,引发了基因剔除技术的一大飞跃,产生了条件性基因剔除(conditional gene knockout)。条件性基因剔除是指某一特定细胞类型或细胞发育的特定阶段剔除某一特定基因的技术。目前多采用Cre-loxP重组系统(Cre-loxP-mediated genereplacement ,Cre-loxP recombination system)。其中Cre重组酶来源于侵染大肠杆菌P1噬菌体。分子量38000,无论在大肠杆菌体内或体外,它都能启动DNA分子间或分子内的联合与重组,重组发生在一个被称为loxP的特异位点(loxp site),且不需要其他的蛋白质因子。在Cre酶存在时,两个loxP位点可以同源重组,切除其中间的部分,留下一个loxP位点。其原理如图23-4所示。研究发现,在哺乳动物细胞中Cre重组酶也同样能引起DNA联合和位点特异性重组。
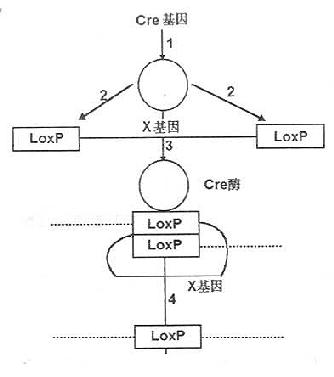
图23-4 Cre酶作用原理
1Cre基因转译成Cre酶;2Cre酶作用于基因组上loxP位点;3Cre酶使两个loxP位点联合;4两个loxP位点发生同源重组切除X基因及一个loxP位点而仅留下一个loxP位点。
Cu等(1994)在鼠的ES细胞中利用Cre-loxP重组系统进行条件性基因剔除的程序如下:①用常规性基因剔除方法将一段新构建的DNA序列整合至内源基因组,这段新构建的DNA序列由新的(突变的)基因片段,选择性标记基因HSV-tk及neor、一个将被取代的正常基因片段等几部分组成。在此新构建的NDA序列中,在可选择性标记基因和正常基因片段的两侧都连有loxP位点;②经过基因剔除的ES细胞被一个编码Cre重组酶载体迅速传染,因此处于两个loxP位点之间的HSV-tk、neor及正常的野生型基因均在Cre酶的作用下而被切除,只在原来的位置上留下了突变的基因和其3’端带有一个loxP位点,其Cre-loxP重组系统进行条件性基因剔除程序如图23-5所示。
条件性基因剔除系统的建立,使得转基因的目的更加明确,效果也进一步精确可靠,是基因剔除方法上的一个重大突破,将给动脉粥样硬化的研究增加更便利的条件。

图23-5 CreloxP重组系统进行条件性基因剔除程序
1有待于被剔除的基因组上某野生型基因片段;2将一个loxP位点插在野生型基因片段的3’端;3将新构建的DNA序列整合到基因组上;4在Cre酶的作用下切除HSV-tk、neor野生型基因及一个loxP位点;5经过Cre-loxP重组系统作用后所形成的DNA序列。
